技术背景
在前面两篇文章中,我们分别介绍了分子动力学模拟软件MindSponge的软件架构和安装与使用。这里我们进入到实用化阶段,假定大家都已经在本地部署好了基于MindSpore的MindSponge的编程环境,开始用MindSponge去做一些真正的分子模拟的工作。那么分子模拟的第一步,我们就需要在MindSponge中去定义一个分子系统Molecule()。
基础类Molecule的解析
我们先来看一下源代码中的Molecule这个类的自我介绍:
class Molecule(Cell):
r"""
Base class for molecular system, used as the "system module" in MindSPONGE.
The `Molecule` Cell can represent a molecule or a system consisting of multiple molecules.
The major components of the `Molecule` Cell is the `Residue` Cell. A `Molecule` Cell can
contain multiple `Residue` Cells.
Args:
atoms(Union[List[Union[str, int]], ndarray]): Array of atoms. The data in array can be str of atom
name or int of atomic number. Defulat: None
atom_name(Union[List[str], ndarray]): Array of atom name with data type `str`. Defulat: None
atom_type(Union[List[str], ndarray]): Array of atom type with data type `str`. Defulat: None
atom_mass(Union[Tensor, ndarray, List[float]]): Array of atom mass of shape `(B, A)` with data type
`float`. Defulat: None
atom_charge(Union[Tensor, ndarray, List[float]]): Array of atom charge of shape `(B, A)` with data type
`float`. Defulat: None
atomic_number(Union[Tensor, ndarray, List[float]]): Array of atomic number of shape `(B, A)` with data type
`int`. Defulat: None
bond(Union[Tensor, ndarray, List[int]]): Array of bond connection of shape `(B, b, 2)` with data
type `int`. Defulat: None
coordinate(Union[Tensor, ndarray, List[float]]): Tensor of atomic coordinates :math:`R` of shape
`(B, A, D)` with data type `float`. Default: None
pbc_box(Union[Tensor, ndarray, List[float]]): Tensor of box size :math:`\vec{L}` of periodic boundary
condition (PBC). The shape of tensor is `(B, D)`,
and the data type is `float`. Default: None
template(Union[dict, str, List[Union[dict, str]]]): Template for molecule. It can be a `dict` in MindSPONGE
template format or a `str` for the filename of a
MindSPONGE template file. If a `str` is given,
it will first look for a file with the same name in the
current directory. If the file does not exist, it will
search in the built-in template directory of
MindSPONGE (`mindsponge.data.template`).
Default: None.
residue(Union[Residue, List[Residue]]): Residue or a list of residues. If template is not None,
only the residues in the template will be used.
Default: None.
length_unit(str): Length unit. If `None` is given, the global length
units will be used. Default: None
Outputs:
- coordinate, Tensor of shape `(B, A, D)`. Data type is float.
- pbc_box, Tensor of shape `(B, D)`. Data type is float.
Supported Platforms:
``Ascend`` ``GPU``
Symbols:
B: Batchsize, i.e. number of walkers in simulation
A: Number of atoms.
b: Number of bonds.
D: Spatial dimension of the simulation system. Usually is 3.
"""
可以先看一下Molecule所接收的信息,其实可以主要分为以下几大类别:
- 原子特征信息。用于区分不同原子之间的差异性,比如atom_name原子名称、atom_type原子类型、atomic_number原子序数等。
- 拓扑信息。在构建Molecule的时候需要传入键连信息bond,否则不带键连关系的Molecule计算出来的力场能量是错误的。
- 构象信息。主要是原子坐标coordinate和周期性边界条件pbc_box,作为近邻表计算和力场能量计算的输入,但不作为拓扑连接信息的输入。
- 模块化信息。除了逐个原子的去构建一个Molecule,还可以定义好一系列完整的残基Residue再输入给Molecule进行构建,或者通过模板template来进行构建。
- 单位信息。主要包含长度单位length_unit和能量单位energy_unit。
上述主要是给Molecule的输入信息,输入给Molecule之后在内部构建build一次,才能得到一个最终的分子系统对象。接下来看看构建之后的Molecule的一些重要内置属性(self.xxx):
- 原子特征信息。除了上述传入的那些信息之外,还有原子数num_atoms,batch数量num_walker以及灵活的维度数量dimension。除了每个原子的基本类型外,还保存了一个heavy_atom_mask重原子的信息,便于快速区分重原子和氢原子。
- 拓扑信息。除了键连关系bonds信息,还有h_bonds氢原子成键的信息。
- 构象信息。主要就是coordinate原子坐标,因为需要在Updater中更新迭代,因此这里的coordinate需要是一个Parameter的类型,而不是普通的Tensor。
- 模块化信息。在构建的过程中,对传入的Residue也都进行了extend,因此最终Residue内部的这些信息,都会被合并到前面提到的Molecule的原子特征信息和拓扑信息、构象信息中,同时会保留一个atom_resid用于追溯原子所在的residue。如果在template模板中有配置一些约束限制,比如settle约束算法相关的参数settle_index和settle_length,也会保存在Molecule的属性中,用于后续约束算法的计算。
- 单位信息。units把相关的单位信息都存储在一个Units对象中,支持从global units中调用,可以随时调用。
除了内置属性,Molecule还有一些内置函数可以关注一下:
- 单位转化。主要是convert_length_from和convert_length_to两个函数,用于执行长度单位的变换。
- 系统扩展函数。比如copy函数,可以用于将本系统拷贝一份,但是该拷贝的过程会生成一个新的对象,而不是原有的Molecule对象。但如果是多个的Molecule对象,可以用内置函数append进行合并。如果需要节省一些麻烦,想对系统进行扩展,可以直接使用内置函数reduplicate,在系统内部复制一份。类似于append的功能,可以使用内置函数add_residue来添加新的residue。上述几种方法主要针对于非周期性的体系,如果是带有周期性边界条件的体系,直接使用repeat_box函数即可完成对体系的快速复制。
- 构建函数。一般情况下对于只是想做MD的童鞋而言,没有必要使用到build_system构建系统和build_space构建构象这些函数,但是如果有需要自行调整Molecule的内容时,就需要重新build一次。
- 补介质函数。一般给定的pdb文件会丢失一些氢原子和溶剂分子的信息,这些都可以在做模拟之前手动补上。目前MindSponge支持的是对体系加水分子fill_water,可以指定溶剂层的厚度,或者指定一个盒子的大小。
- 回调函数。在深度学习或者MindSponge分子动力学模拟的过程中,我们会使用到回调函数CallBack来对输出内容进行追踪。但是CallBack本身是不保存任何体系相关的信息的,因此追踪的内容其实也是从Molecule和ForceField内部进行回调。比如在Molecule中,可以调用get_atoms,get_coordinate,get_pbc_box等等函数,而如果直接使用MindSpore的Cell中所特有的construct函数,这里也会返回coordinate和pbc_box两个信息,这些都可以认为是Molecule类的“回调函数”。
从模板定义一个分子
关于MindSponge的安装和使用,在这里我们就不重复赘述了,假设大家已经完成了MindSponge的安装。但是需要提一句的是,在开始MindSponge模拟前,最好在python脚本的最前面加上这样一些环境变量的配置,否则容易报错:
import os
os.environ['GLOG_v']='4'
os.environ['MS_JIT_MODULES']='sponge'
接下来我们就可以简单的使用模板文件去创建一个新的分子:
from sponge import Molecule
system = Molecule(template='water.spce.yaml')
print ('The number of atoms in the system is: ', system.num_atoms)
print ('All the atom names in the system are: ', system.atom_name)
print ('The coordinates of atoms are: \n{}'.format(system.coordinate.asnumpy()))
输出的结果如下所示:
The number of atoms in the system is: 3
All the atom names in the system are: [['O' 'H1' 'H2']]
The coordinates of atoms are:
[[[ 0. 0. 0. ]
[ 0.08164904 0.0577359 0. ]
[-0.08164904 0.0577359 0. ]]]
这里因为water.spce.yaml是一个预置的模板,类似的还有water.tip3p.yaml。这种预置的模板我们可以直接当做template来创建,但如果是用户自行定义的模板文件,最好在这里写清楚yaml文件的绝对路径,否则会导致报错。相关yaml文件的内容如下所示:
template:
base: water_3p.yaml
WAT:
atom_mass: [15.9994, 1.008, 1.008]
atom_charge: [-0.8476, 0.4238, 0.4238]
settle:
mandatory: false
length_unit: nm
distance:
OW-HW: 0.1
HW-HW: 0.16330
molecule:
residue:
- WAT
length_unit: nm
coordinate:
- [0.0, 0.0, 0.0]
- [0.081649043, 0.057735897, 0.0]
- [-0.081649043, 0.057735897, 0.0]
这里的base是指向了另外一个较为基础的yaml参数文件:
template:
WAT:
atom_name: [O, H1, H2]
atom_type: [OW, HW, HW]
atom_mass: [16.00, 1.008, 1.008]
atomic_number: [8, 1, 1]
bond:
- [0, 1]
- [0, 2]
head_atom: null
tail_atom: null
有了这些参考,用户就可以自行定义一些模板,用于计算。
从文件定义一个分子
MindSponge也支持一些特定格式的分子导入,比如mol2格式的分子和pdb格式的蛋白质分子,这个章节介绍一下如何将文件导入为一个MindSponge的Molecule。比如我这里有一个非常简单的pdb格式的多肽链:
REMARK Generated By Xponge (Molecule)
ATOM 1 N ALA 1 -0.095 -11.436 -0.780
ATOM 2 CA ALA 1 -0.171 -10.015 -0.507
ATOM 3 CB ALA 1 1.201 -9.359 -0.628
ATOM 4 C ALA 1 -1.107 -9.319 -1.485
ATOM 5 O ALA 1 -1.682 -9.960 -2.362
ATOM 6 N ARG 2 -1.303 -8.037 -1.397
ATOM 7 CA ARG 2 -2.194 -7.375 -2.328
ATOM 8 CB ARG 2 -3.606 -7.943 -2.235
ATOM 9 CG ARG 2 -4.510 -7.221 -3.228
ATOM 10 CD ARG 2 -5.923 -7.789 -3.136
ATOM 11 NE ARG 2 -6.831 -7.111 -4.087
ATOM 12 CZ ARG 2 -8.119 -7.421 -4.205
ATOM 13 NH1 ARG 2 -8.686 -8.371 -3.468
ATOM 14 NH2 ARG 2 -8.844 -6.747 -5.093
ATOM 15 C ARG 2 -2.273 -5.882 -2.042
ATOM 16 O ARG 2 -1.630 -5.388 -1.119
ATOM 17 N ALA 3 -3.027 -5.119 -2.777
ATOM 18 CA ALA 3 -3.103 -3.697 -2.505
ATOM 19 CB ALA 3 -1.731 -3.041 -2.625
ATOM 20 C ALA 3 -4.039 -3.001 -3.483
ATOM 21 O ALA 3 -4.614 -3.643 -4.359
ATOM 22 N ALA 4 -4.235 -1.719 -3.394
ATOM 23 CA ALA 4 -5.126 -1.057 -4.325
ATOM 24 CB ALA 4 -6.538 -1.625 -4.233
ATOM 25 C ALA 4 -5.205 0.436 -4.039
ATOM 26 O ALA 4 -4.561 0.930 -3.116
ATOM 27 OXT ALA 4 -5.915 1.166 -4.728
TER
使用MindSponge来读取该pdb文件的方法为[*注:由于一般pdb文件中会忽略氢原子,因此加载的时候需要使用rebuild_hydrogen将其重构成一个完整的分子]:
from sponge import Protein
system = Protein('case1.pdb', rebuild_hydrogen=True)
print ('The number of atoms in the system is: ', system.num_atoms)
print ('All the atom names in the system are: ', system.atom_name)
相应的输出结果为:
[MindSPONGE] Adding 57 hydrogen atoms for the protein molecule in 0.007 seconds.
The number of atoms in the system is: 57
All the atom names in the system are: [['N' 'CA' 'CB' 'C' 'O' 'H1' 'H2' 'H3' 'HA' 'HB1' 'HB2' 'HB3' 'N' 'CA'
'CB' 'CG' 'CD' 'NE' 'CZ' 'NH1' 'NH2' 'C' 'O' 'H' 'HA' 'HB2' 'HB3' 'HG2'
'HG3' 'HD2' 'HD3' 'HE' 'HH11' 'HH12' 'HH21' 'HH22' 'N' 'CA' 'CB' 'C'
'O' 'H' 'HA' 'HB1' 'HB2' 'HB3' 'N' 'CA' 'CB' 'C' 'O' 'OXT' 'H' 'HA'
'HB1' 'HB2' 'HB3']]
可以看到的是,在对应的位置上,我们将氢原子补在了一个相对合适的位置。一般情况下,重构完氢原子之后,需要对系统进行一个能量极小化,否则会导致初始系统的能量过于不稳定。具体的加氢效果可以看一下这个运行的结果:
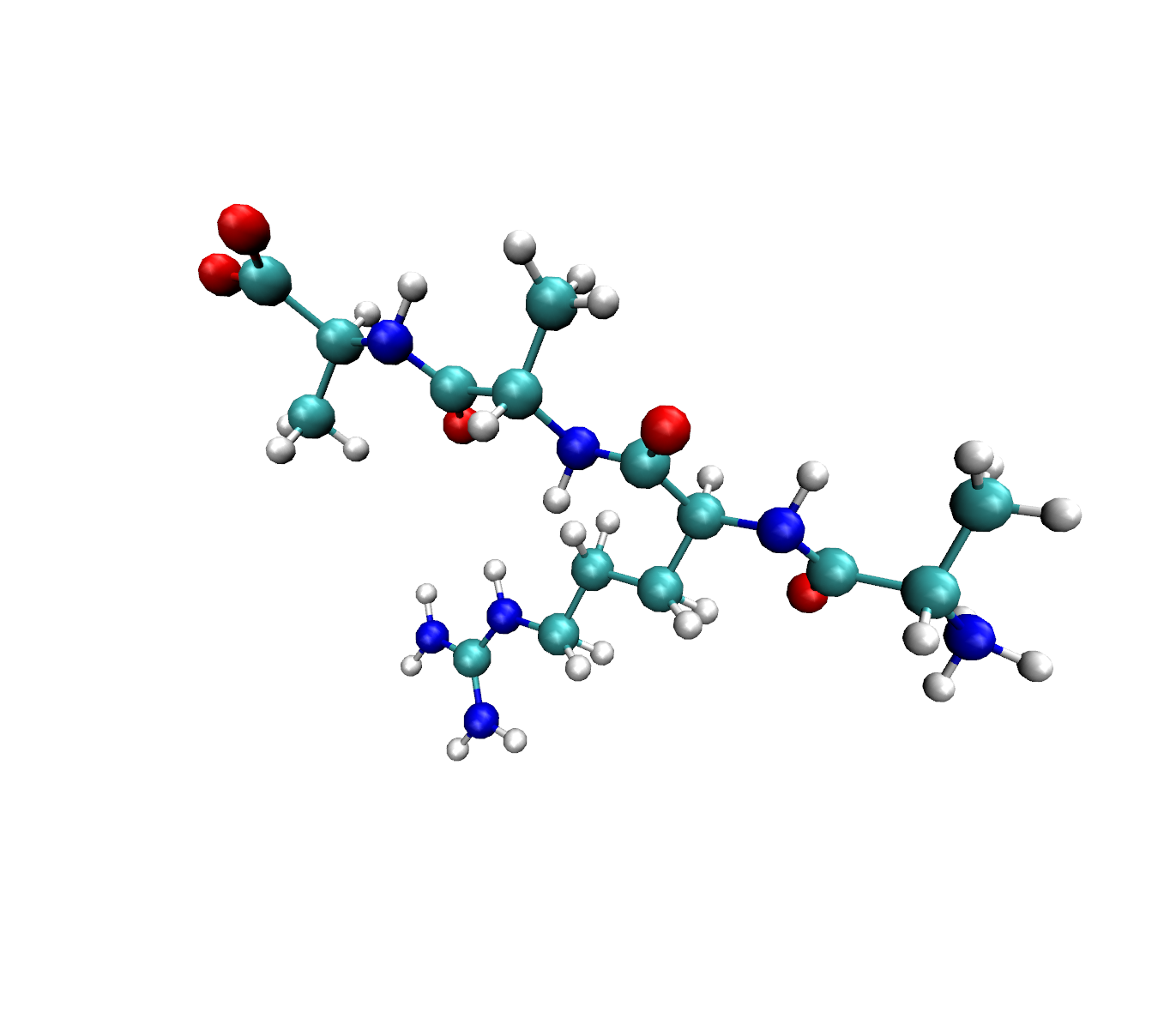
其实加氢是很难做到一步到位的,但是我们可以尽可能的将氢原子摆放在一个相对合理的位置,便于后续的能量计算和优化。
自定义分子
由于python这一编程语言的灵活性,使得我们不仅支持从文件和模板文件中去定义一个分子系统,还可以直接用脚本的形式传一系列的python列表给Molecule来构建一个分子系统。比如我们只传原子类型和坐标还有键连关系,就能构建一个简单的水分子:
from sponge import Molecule
system = Molecule(atoms=['O', 'H', 'H'],
coordinate=[[0, 0, 0], [0.1, 0, 0], [-0.0333, 0.0943, 0]],
bonds=[[[0, 1], [0, 2]]])
print ('The number of atoms in the system is: ', system.num_atoms)
print ('All the atom names in the system are: ', system.atom_name)
print ('The coordinates of atoms are: \n{}'.format(system.coordinate.asnumpy()))
相应的输出结果如下所示:
The number of atoms in the system is: 3
All the atom names in the system are: [['O' 'H' 'H']]
The coordinates of atoms are:
[[[ 0. 0. 0. ]
[ 0.1 0. 0. ]
[-0.0333 0.0943 0. ]]]
总结概要
本文通过解析MindSponge的源码实现,详细介绍了在MindSponge中Molecule基础分子类的内置属性和内置函数,以及三种相应的分子系统定义方法:我们既可以使用yaml模板文件来定义一个分子系统,也可以从mol2和pdb文件格式中直接加载一个Molecule,还可以直接使用python列表的形式传入一些手动定义的内容,直接构建一个Molecule。有了最基础的分子系统之后,后面就可以开始定义一些能量项和迭代器,开始分子动力学模拟。